Taller Teórico-Práctico de Metabarcoding 2024
Tutorial de Metabarcoding
Tabla de Contenidos
- Introducción
- Sobre los datos que vamos a manejar
- Paso por paso
- Conclusiones
Introducción
El metabarcoding es una técnica de secuenciación de ADN que permite identificar y caracterizar la biodiversidad presente en una muestra ambiental. Utiliza secuencias de ADN seleccionadas para determinar rápidamente las especies que componen una comunidad biológica. Estas secuencias normalmente son marcadores taxonómicos con características específicas ya seleccionadas. Para el caso del taller de hoy, ese marcador será el 16S, por características que estaremos definiendo después.
Asumimos que los ácidos nucléicos y las proteínas cambian con el tiempo. Es por ello que pueden considerarse como registros de la historia evolutiva. Teoretizando que estos cambios se dan al azar y que aumentan de manera lineal, se deduce que estos cambios pueden reflejar la distancia evolutiva existente entre ellas.
Los ribosomas son orgánulos que utilizan los organismos para la síntesis de proteínas. El ribosoma bacteriano tiene un índice de sedimentación de 70S (coeficiente de Svedberg), razón por la cuál se presenta usualmente con esta unidad. Puede disociarse en dos subunidades: La subunidad grande 50S y la subunidad pequeña 30S.
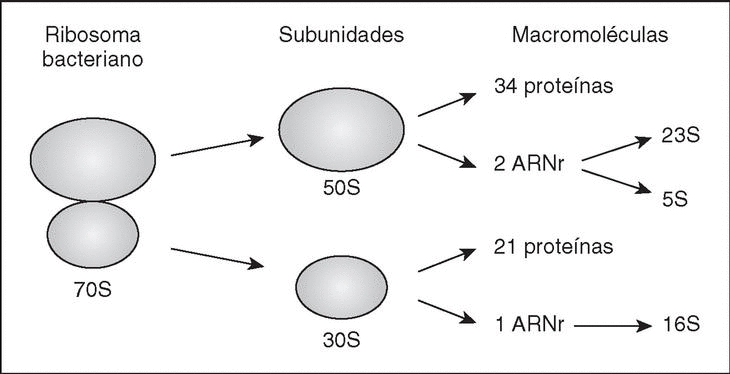
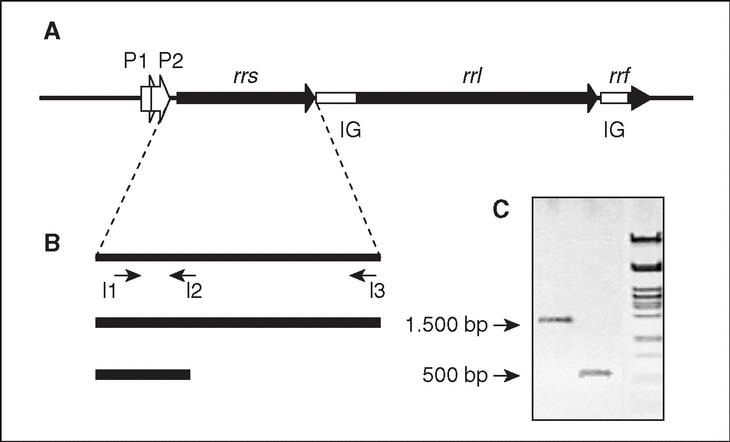
Los genes que codifican los ARN ribosomales están organizados en operones (conjunto de genes que se transcriben a partir de la misma región promotora). Cada operón cuenta con regiones rrl (que codifica para 23S), rrs (16S) y 5S (rrf). Las regiones promotoras P1 y P2 se sitúanen la región anterior a rrs. Las regiones se encuentran separadas por regiones intergénicoas. Al momento que la ARNasaIII procesa el producto del operón mediante cortes, separa las tres clases de ARNr y las dos IG (1).
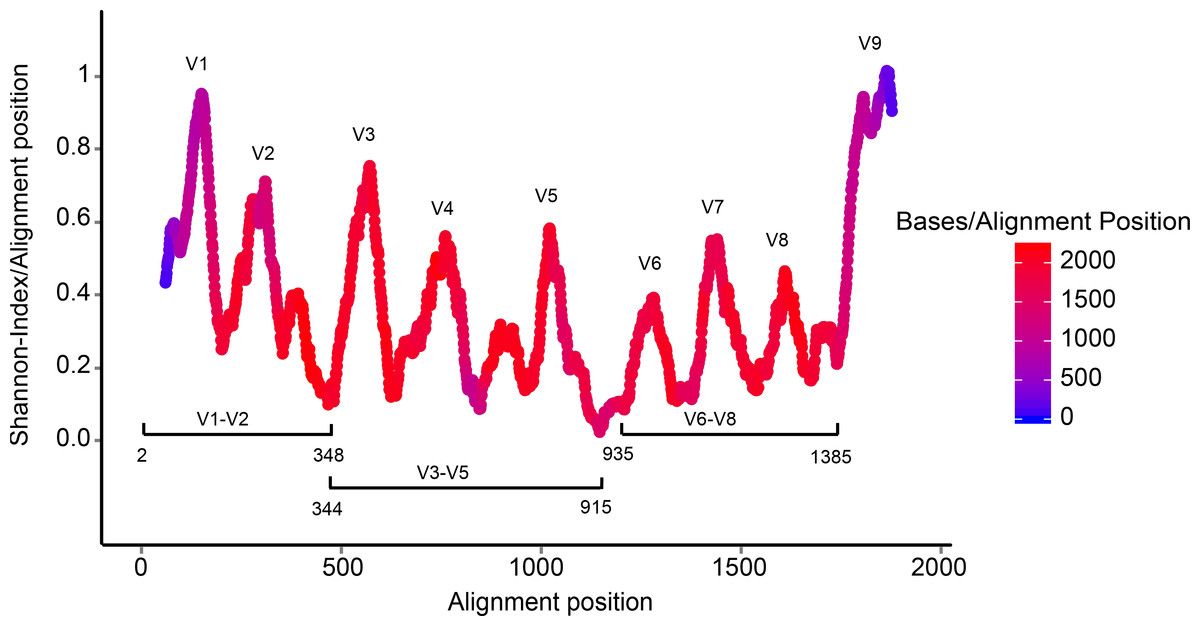
¿Por qué 16S?
El gen de la secuencia ribosomal 16S presenta ciertas características únicas que lo hacen ideal para el trabajo (2):
- Presente en todos los organismos vivos.
- No presenta recombinación, solo es una copia.
- Altamente conservado y con regiones que son altamente variables (raro, pero básicamente lo primero indica que su función se mantiene porque es vital, mientras que sus regiones varían)
- Gran base de datos de referencia.
Sobre los datos que vamos a manejar
Para este tutorial, estaremos realizando lo que se describe en este tutorial de Galaxy. El procedimiento será similar, pero estaré presentando esta información en español y con actualizaciones respecto a las imagenes utilizadas. Así que todos los créditos van a ellos, realmente. Mayor detalle puede ser observado en su página.
Estaremos utilizando data del laboratorio Schloss. Estos describen su experimento de la siguiente manera:
“El laboratorio Schloss está interesado en comprender el efecto de la variación normal en el microbioma intestinal en la salud del huésped. Con ese fin, recolectamos heces frescas de ratones diariamente durante 365 días después del destete. Durante los primeros 150 días después del destete (dpw), no se hizo nada con nuestros ratones, excepto permitirles comer, engordar y estar felices. Nos preguntábamos si el cambio rápido de peso observado durante los primeros 10 dpw afectó la estabilidad del microbioma en comparación con el microbioma observado entre los días 140 y 150. Analizaremos un solo ratón en 20 puntos de tiempo diferentes (10 temprano, 10 tardío). Para evaluar la tasa de error del análisis y la configuración experimental, el laboratorio Schloss también secuenció una comunidad simulada con una composición conocida (ADN genómico de 21 cepas bacterianas).”
Paso por paso
Crear cuenta en Galaxy e importar secuencias
Primero es necesario generar una cuenta en Galaxy. Utilizaremos esta plataforma puesto que no requiere ninguna instalación complicada y nos permite centrarnos en los pasos. Para ello requieres hacer lo siguiente:

Luego de este paso, nos toca importar la data a utilizar. Para ello utilizaremos una forma de importación especial. Requeriremos URLs de nuestras muestras. Aquí van:
https://zenodo.org/record/800651/files/F3D0_R1.fastq
https://zenodo.org/record/800651/files/F3D0_R2.fastq
https://zenodo.org/record/800651/files/F3D141_R1.fastq
https://zenodo.org/record/800651/files/F3D141_R2.fastq
https://zenodo.org/record/800651/files/F3D142_R1.fastq
https://zenodo.org/record/800651/files/F3D142_R2.fastq
https://zenodo.org/record/800651/files/F3D143_R1.fastq
https://zenodo.org/record/800651/files/F3D143_R2.fastq
https://zenodo.org/record/800651/files/F3D144_R1.fastq
https://zenodo.org/record/800651/files/F3D144_R2.fastq
https://zenodo.org/record/800651/files/F3D145_R1.fastq
https://zenodo.org/record/800651/files/F3D145_R2.fastq
https://zenodo.org/record/800651/files/F3D146_R1.fastq
https://zenodo.org/record/800651/files/F3D146_R2.fastq
https://zenodo.org/record/800651/files/F3D147_R1.fastq
https://zenodo.org/record/800651/files/F3D147_R2.fastq
https://zenodo.org/record/800651/files/F3D148_R1.fastq
https://zenodo.org/record/800651/files/F3D148_R2.fastq
https://zenodo.org/record/800651/files/F3D149_R1.fastq
https://zenodo.org/record/800651/files/F3D149_R2.fastq
https://zenodo.org/record/800651/files/F3D150_R1.fastq
https://zenodo.org/record/800651/files/F3D150_R2.fastq
https://zenodo.org/record/800651/files/F3D1_R1.fastq
https://zenodo.org/record/800651/files/F3D1_R2.fastq
https://zenodo.org/record/800651/files/F3D2_R1.fastq
https://zenodo.org/record/800651/files/F3D2_R2.fastq
https://zenodo.org/record/800651/files/F3D3_R1.fastq
https://zenodo.org/record/800651/files/F3D3_R2.fastq
https://zenodo.org/record/800651/files/F3D5_R1.fastq
https://zenodo.org/record/800651/files/F3D5_R2.fastq
https://zenodo.org/record/800651/files/F3D6_R1.fastq
https://zenodo.org/record/800651/files/F3D6_R2.fastq
https://zenodo.org/record/800651/files/F3D7_R1.fastq
https://zenodo.org/record/800651/files/F3D7_R2.fastq
https://zenodo.org/record/800651/files/F3D8_R1.fastq
https://zenodo.org/record/800651/files/F3D8_R2.fastq
https://zenodo.org/record/800651/files/F3D9_R1.fastq
https://zenodo.org/record/800651/files/F3D9_R2.fastq
https://zenodo.org/record/800651/files/Mock_R1.fastq
https://zenodo.org/record/800651/files/Mock_R2.fastq
A continuación, importamos también la data complementaria:
https://zenodo.org/record/800651/files/HMP_MOCK.v35.fasta
https://zenodo.org/record/800651/files/silva.v4.fasta
https://zenodo.org/record/800651/files/trainset9_032012.pds.fasta
https://zenodo.org/record/800651/files/trainset9_032012.pds.tax
https://zenodo.org/record/800651/files/mouse.dpw.metadata
Proseguiremos creando un dataset. Para ello, seleccionaremos todas nuestras secuencias con un check y le daremos a “Build dataset pairs list”. Automáticamente, Galaxy deducirá que tienes secuencias forward (R1) y reverse (R2). De no ser así, se le puede agregar ese filtro de todos modos. Vemos que Galaxy también le ha asignado un nombre a cada uno de los archivos pareados, de acuerdo al nombre que queda detrás del guión bajo.

Por último, agrega el nombre de tu preferencia en la parte de abajo.
Control de calidad
Antes de realizar el control de calidad de nuestras secuencias, procederemos a crear contigs en base a nuestros archivos. Esto se logra utilizando la herramienta Make.contig disponible en Galaxy.
Realizaremos, entonces, los siguientes pasos:
- Filtrar por longitud: Sabemos que nuestra región tiene 240 bp. Las secuencias que sean más largas son en realidad malos ensamblajes.
- Remover contigs de baja calidad: Cualquier secuencia que tenga baja calidad entrará aquí.
- Deduplicar secuencias idénticas: Sabemos que tenemos varias secuencias que son básicamente el mismo organismo. Entonces, escogeremos solo las secuencias únicas.
- Contar secuencias: Un procedimiento que permite saber el número de secuencias en una muestra.
Para acelear el procedimiento, utilizaremos un workflow que se ejecutará de la siguiente manera:

Si entramos a la opción del Workflow completo, podremos ver que la opción para filtrar secuencias se encuentra en Screen.seqs, en dónde hay una longitud máxima ya establecida.
Es importante notar que, si bien trabajaremos con secuencias únicas, nuestra tabla de conteo mantiene los números originales, y que lo que calculemos como único es solo una representación de lo que realmente tenemos.
Una vez que terminamos con ese procedimiento, pasamos a identificar nuestras secuencias.
Alineamiento con base de datos

Esto se realizará con la herramienta align.Seqs, que nos permitirá alinear nuestras secuencias con la base de datos disponible para nuestra muestra (en este caso, Silva). Para ello, colocaremos las siguientes opciones:
Más limpieza y eliminación de quimeras
Logramos determinar las secuencias que se alinean con nuestra BD. Sin embargo, aún nos quedamos con secuencias no alineadas, y por ahí tenemos secuencias híbridas artificiales (productos normales en PCRs) que no se alinean tampoco. Estas secuencias se conocen como quimeras, como las criaturas mitológicas griegas que combinaban varios animales en una sola. Utilizaremos el siguiente workflow, que nos permitirá hacer varios pasos de limpieza de un porrazo.

Hay que prestarle atención a los datos que nos otorgan las herramientas respecto a las secuencias removidas y la cantidad de secuencias que tenemos.
Asignación de taxonomía y remoción de secuencias no bacterianas.
Ahora si procederemos a asignar una taxonomía. Para ello, haremos la identificación en base a nuestra BD. Utilizaremos nuevamente un workflow de Galaxy con los siguientes parámetros:

A través de este paso, podemos determinar los grupos taxonómicos presentes en nuestras muestras.
Clustering por OTUs
Los análisis de 16S normalmente trabajan a partir de OTUs (Operational Taxonomic Unit). Esta es una clusterización realizada computacionalmente, es decir, a partir de la similitud entre secuencias. Y, eso quiere decir, que no tienen una significancia biológica en sí. Por el momento solo una clusterización cruda y dura a partir de nuestros datos.
Para ello, utilizaremos el siguiente workflow de la siguiente manera:

Visualización
Por último, realizaremos la visualización de nuestras muestras utilizando Taxonomy-to-Krona y Krona-pie-chart. Esto lo realizaremos con los siguientes parámetros:
-
Taxonomy-to-Krona: Se añade el output Taxonomy deClassify.out -
Krona pie chart: Se añade el output Tabular deTaxonomy-to-Krona.
Esto se añade de la siguiente manera:

Con lo que quedaría lo siguiente:

Conclusión
El metabarcoding 16S es una herramienta poderosa para estudiar comunidades microbianas a través de la secuenciación del gen ARNr 16S. Sin embargo, presenta algunas limitaciones importantes. En primer lugar, la asignación taxonómica basada en secuencias de 16S depende de bases de datos de referencia, y la calidad y exhaustividad de estas bases de datos pueden variar. Por lo tanto, los resultados deben interpretarse con precaución, especialmente cuando se trata de taxas poco conocidas o no cultivadas. A pesar de estas limitaciones, el metabarcoding 16S sigue siendo una herramienta valiosa para explorar la diversidad microbiana en diferentes entornos y proporcionar información clave para la investigación ambiental y la salud pública.
Enjoy Reading This Article?
Here are some more articles you might like to read next: